产品货号:
YTB4039
中文名称:
WELQut蛋白酶
英文名称:
WELQut Protease,5U/μl
产品规格:
500U|2500U|10kU
发货周期:
1~3天
产品价格:
询价
本制品是表达纯化获得的重组蛋白酶。WELQut蛋白酶能特异性地识别四肽序列Trp-Glu-Leu-Gln (W-E-L-Q),并对Gln (Q)之后的肽键进行酶切,常用于重组蛋白除去His或者其它融合标签,其最突出优势是酶切后不会在目的蛋白的N端留下任何额外的氨基酸残基。

本WELQut蛋白酶反应条件比较宽泛。WELQut蛋白酶是一种源自金黄色葡萄球菌的丝氨酸蛋白酶类似蛋白,该蛋白酶反应时不需要特殊的反应缓冲液,能在比较宽泛的温度区间(4~30℃)及pH区间(6.5-9.0)识别WELQ多肽序列,并在Gln (Q)之后进行蛋白或多肽的剪切。
本WELQut蛋白酶能适用于His标签蛋白的柱上酶切。本WELQut蛋白酶带有His标签,特别适用于His标签蛋白的柱上酶切。在切割His标签蛋白的时候,切下的His标签和WELQut蛋白酶可结合于His纯化柱上,而目的蛋白在穿透液中,这样洗脱下来的蛋白中理论上就不会含有His标签和WELQut蛋白酶,极大地方便了目的蛋白的纯化。如果不进行柱上酶切,分别过镍柱和能除去目的蛋白融合标签的纯化柱也可以方便地去除WELQut蛋白酶和目的蛋白上切割下来的标签。

| 组分 | 500U | 2500U | 10kU |
| WELQut蛋白酶(5U/μL) | 100μL | 500μL | 2mL |
| 说明书 | 1份 | ||
保存:-20℃,有效期2年。

10mM Na2HPO4,1.8mM KH2PO4,140mM NaCl,2.7mM KCl,50% (v/v) Glycerol,pH7.3。

WELQut Protease的酶切体系中可以兼容:0.01~1% Triton X-100/Tween-80,5~50mM DTT,100mM NaCl,20~300mM Imidazole,100mM Tris-HCl (pH8.0),100mM Na3PO4 (pH7.4),20mM K3PO4 (pH7.4)。

WELQut Protease分子量大小约22kDa,纯度≥95%。不含DNA内切酶和外切酶,不含RNA酶,不含磷酸酯酶。

2M尿素、0.25M盐酸胍会抑制WELQut Protease的部分酶活性。

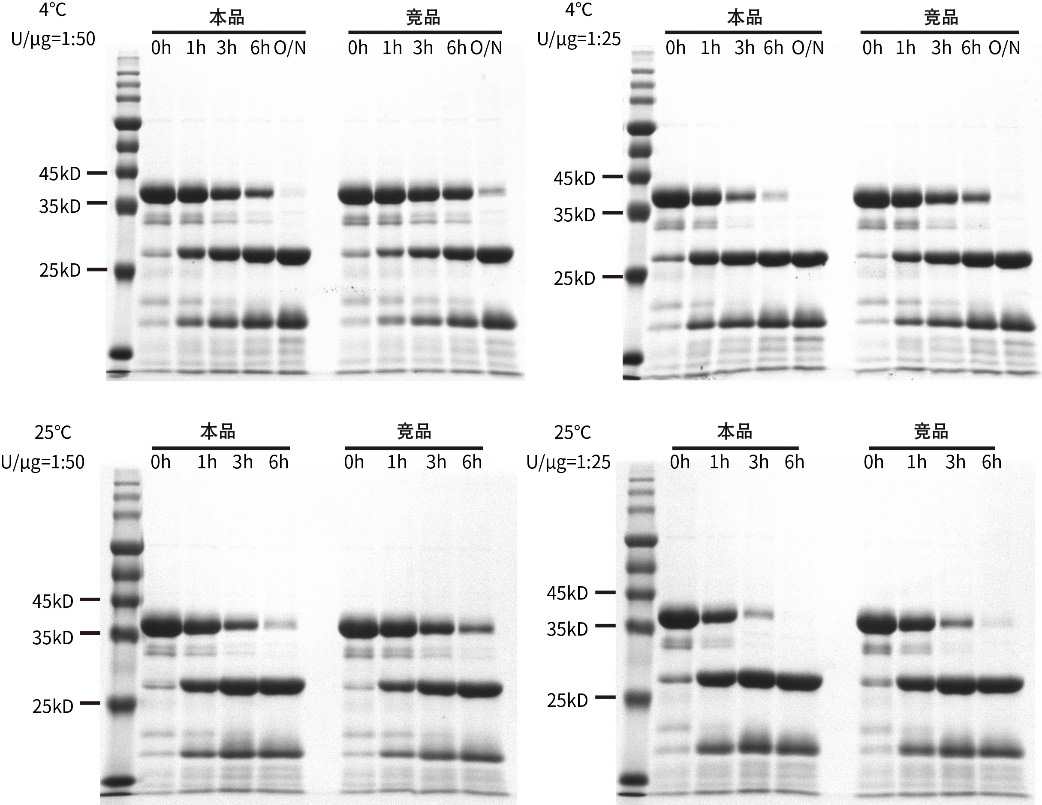
图1.我司的WELQut蛋白酶与T公司同类产品切割GST标签融合蛋白(WELQ四氨基酸序列位于GST和10kD目的蛋白之间)的效果图。将含有WELQut Protease识别位点的36kD GST标签融合蛋白与WELQut Protease进行反应,底物的用量为50μg,酶的用量分别为1U和2U,在50mM Tris-HCl (pH8.0)的缓冲液中反应,设置4℃与25℃两种反应温度,在反应0、1、3、6小时以及过夜(Overnight,O/N) (超过16小时)后取样,进行SDS-PAGE电泳和考马斯亮蓝染色。酶切产物大小为约10kD的目的蛋白和约26kD的GST标签。如图所示,本制品与T公司的竞品具有相当的活性。实际效果会因样品种类、检测条件等不同而存在差异,本图仅供参考。

- WELQut Protease酶切条件优化:
WELQut Protease识别位点的暴露程度、位点附近的氨基酸序列以及蛋白的聚集程度都会影响WELQut Protease的酶切效果。因而对不同的蛋白进行酶切时,建议通过预实验来优化酶切条件。建议从以下三个方面进行酶切条件的优化:反应温度(4~30℃)、反应时间(1-12h或过夜)、酶的用量(酶活性单位数量与目的蛋白μg数比例(U/μg)为1:5~1:100)。如下是一个简单的摸索酶用量、反应时间的实验方案。a.按照下表设置酶切反应体系:成分 用量 Target Protein 50μg WELQut蛋白酶(5U/μl) 0.5/1/2/10U Reaction Buffer To 50μL - 设置酶与底物蛋白的比例分别为1:10、1:50、1:25、1:5(U/μg)。除1:5的比例外,建议将WELQut Protease稀释至0.5U/μl后使用。
- 建议使用10~100mM Tris-HCl (pH8.0)作为Reaction Buffer。
- 设置酶与底物蛋白的比例分别为1:10、1:50、1:25、1:5(U/μg)。除1:5的比例外,建议将WELQut Protease稀释至0.5U/μl后使用。
- 将反应混合物置于15~30℃反应。分别在反应1、3、6、12小时(或过夜)后,取出10μL酶切产物,用于后续的SDS-PAGE检测。当反应时长大于16小时时,建议酶切反应温度设置在20℃以下。
- 将上一步骤的留取的蛋白样品进行SDS-PAGE电泳分析,选择比较理想的反应的条件。确定了优化后的反应条件后,后续可以等比例扩大反应体系,选择进行溶液体系中的酶切或柱上酶切。如果用于柱上酶切,可能后续还需要进一步的调整酶用量等的酶切条件优化。
- 融合蛋白在溶液体系中的酶切和纯化:
- 推荐使用10~100mM Tris-HCl (pH8.0)、10~100mM Na3PO4 (pH7.4)或10~20mM K3PO4 (pH7.4)作为Reaction Buffer进行酶切。
- 如果酶切后的目的蛋白后续需要进行亲和层析,Reaction Buffer可以含有50mM NaCl以及5~20mM Imidazole。
- 按照优化后的酶与目的蛋白的比例和酶切时间,加入WELQut Protease,在15~30℃中进行反应。
- 如果蛋白不稳定或者酶切反应时长大于16小时,建议选用4~20℃进行反应。
- 将酶切后的蛋白样品加入预先用Reaction Buffer平衡好的GoldBalb His-tag Purification Resin (货号:YT616、YTB4204)中,室温结合20~30分钟。
- 500×g离心5分钟,收集上清,其中含有切除了标签的目的蛋白,WELQut Protease则结合在凝胶沉淀中。如果目的蛋白是His标签蛋白,那么残留的没有被酶切的His标签蛋白、WELQut Protease和酶切下来的His标签则结合在凝胶沉淀中,切除了标签的目的蛋白在溶液中。如果目的蛋白不是His标签融合蛋白,那么切除下来的带有WELQ的片段,还需要用适当的方法去除,例如如果是GST标签,那么可使用GST纯化柱去除GST标签。
- 推荐使用10~100mM Tris-HCl (pH8.0)、10~100mM Na3PO4 (pH7.4)或10~20mM K3PO4 (pH7.4)作为Reaction Buffer进行酶切。
- 融合蛋白的柱上酶切和纯化:
如果目的蛋白带有His标签并且His标签的羧基端(C端)同时带有WELQ序列,本WELQut Protease可以在镍柱亲和层析过程中进行融合蛋白的酶切。目的蛋白在与镍柱结合时被切割,并进一步被洗脱,留下目的蛋白的His标签和带有His标签的WELQut Protease于柱上。- 利用SDS-PAGE和考马斯亮蓝染色估计待纯化和酶切的目的蛋白的总量。
- 按照常规操作流程将目的蛋白结合至镍柱上。
- 使用2~4倍柱体积缓冲液,如50~100mM Tris-HCl,50mM NaCl,5~20mM Imidazole (pH8.0)、50~100mM Na3PO4,50mM NaCl,5~20mM Imidazole (pH7.4),或者10~20mM K3PO4,50mM NaCl,5~20mM Imidazole (pH7.4),平衡亲和层析柱2次。
- 按照酶切优化实验中确定的蛋白酶/重组蛋白比例,将WELQut Protease加入上一步镍柱平衡用的缓冲液中混匀后,加到柱上。
- 如果蛋白酶/重组蛋白比例尚未确定,推荐使用1:20(U/μg)或者1:10(U/μg)的比例。
- 在15~30℃中反应适当时间。
- 如果反应时长尚未确定,推荐在4~20℃中反应12小时或者过夜。
- 收集洗脱下来的目的蛋白。亲和标签和带有His标签的WELQut Protease结合于柱上。
- 利用SDS-PAGE和考马斯亮蓝染色估计待纯化和酶切的目的蛋白的总量。

- 为什么酶切不完全?
- 可能是酶与底物蛋白的比例不合适。建议确定待酶切目的蛋白的含量,调整加入WELQut蛋白酶的量。
- 可能是孵育时间不够长。建议延长反应时间。
- 可能是酶切位点并未表达或发生了突变。建议测序确认序列是否正确。
- 可能是酶切位点并未暴露出来。建议尝试使用非离子型变性剂(如0.01~1% Triton X-100/Tween-80)使蛋白发生可逆变性,从而将酶切位点暴露出来。
- 可能是缓冲液内含有WELQut蛋白酶的抑制剂。建议将目的蛋白透析至合适的反应缓冲液中。
- 可能是酶与底物蛋白的比例不合适。建议确定待酶切目的蛋白的含量,调整加入WELQut蛋白酶的量。
- 为什么会出现不正确的条带?
- 可能是目的蛋白上出现了其它类似的酶切位点。建议调整酶切条件(如盐浓度、反应时间或者温度),降低类似酶切位点的暴露程度。如果待表达纯化的目的蛋白本身含有WELQ序列,就不适合使用WELQut蛋白酶进行酶切了。
- 可能是蛋白在菌体内发生降解。建议使用蛋白酶缺陷的菌株。
- 可能是目的蛋白上出现了其它类似的酶切位点。建议调整酶切条件(如盐浓度、反应时间或者温度),降低类似酶切位点的暴露程度。如果待表达纯化的目的蛋白本身含有WELQ序列,就不适合使用WELQut蛋白酶进行酶切了。
相关搜索:WELQut蛋白酶,His标签,GST标签,融合蛋白,标签去除,Trp-Glu-Leu-Gln,W-E-L-Q,WELQut Protease,5U/μl



